Genes y factores ambientales en el desarrollo de asma y atopia
Click here to load reader
Transcript of Genes y factores ambientales en el desarrollo de asma y atopia

REVISIÓN
Genes y factores ambientales en el desarrollo de asma y atopia
María José Torres Galván y José Carlos Rodríguez Pérez
Unidad de Investigación. Hospital de Gran Canarias Dr. Negrín. Las Palmas de Gran Canaria.
Genes; Factores ambientales; Asma; Atopia
6
Las enfermedades alérgicas afectan de un modo u otro agran parte de la humanidad y son una fuente importante dediscapacidad en el mundo occidental. Su prevalencia va enaumento y ha alcanzado proporciones epidémicas. El asma,una de las enfermedades alérgicas más graves, afecta a155 millones de personas en el mundo; en países comoGran Bretaña y Australia, uno de cada 4 menores de 14años es asmático1. El coste de su tratamiento es elevado: 6 billones de dólares al año en los Estados Unidos; 3 y 1,6billones de dólares al año en Alemania y Gran Bretaña, res-pectivamente. Más de la mitad de este gasto es hospitalario,y el 80% es atribuible a sólo un 20% de los pacientes. Elmercado de la industria farmacéutica relativo al asma es de5,5 billones de dólares al año2.
Alergia y atopia
El término «alergia» se creó con el significado de «desvia-ción del estado original» cuando comenzó a observarse quela inyección de proteínas y suero daba lugar a reaccionesinmunológicas dañinas3. La palabra «atopia» fue acuñadapara diferenciar el síndrome familiar de asma y fiebre delheno relacionado con estas reacciones4. Actualmente, am-bos términos se utilizan como sinónimos.La atopia se define generalmente como una condición ca-racterizada por la respuesta IgE exagerada frente a antíge-nos inocuos para la mayoría de las personas, asociada amenudo con reacciones alérgicas respiratorias (rinitis, asmabronquial y alveolitis alérgica), cutáneas (urticaria, eccema ydermatitis de contacto) y oculares (conjuntivitis). Los indivi-duos atópicos suelen presentar títulos elevados de IgE total,así como IgE específica y reactividad cutánea a alergenoscomunes como pólenes, ácaros presentes en el polvo decasa (los más importantes en el caso del asma), mohos, de-rivados epidérmicos de animales o alimentos. La exposiciónanual de un individuo a los alergenos suele ser del orden demicrogramos.Los alergenos, que parecen inofensivos por sí mismos, cau-san enfermedad debido a la intensidad de la respuesta queprovocan en el sistema inmunológico. El mecanismo desensibilización a los alergenos de los individuos atópicos y lacascada inmunológica desencadenada tras la exposición re-petida a los mismos no se conocen completamente. Noobstante, sabemos que se trata de un proceso fisiológicocomplejo, que implica interacciones e intercambio de seña-les entre distintos tipos celulares, mediadores de inflama-ción y mecanismos neurológicos.
Correspondencia: Dr. J.C. Rodríguez Pérez.Unidad de Investigación. Hospital de Gran Canarias Dr. Negrín.Barranco de la Ballena, s/n. 35020 Las Palmas de Gran Canaria.Correo electrónico: [email protected]
Recibido el 14-2-2000; aceptado para su publicación el 16-3-2000
Med Clin (Barc) 2000; 114: 698-703
98
Mecanismo de la reacción alérgica
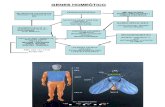
La sintomatología alérgica está determinada por la libera-ción de sustancias vasoactivas por parte de mastocitos y ba-sófilos, al interaccionar el alergeno con las IgE fijadas a sumembrana. En la fisiopatología de la reacción atópica sedistinguen dos fases: una inmediata, desencadenada por lainteracción IgE-alergeno, que libera los mediadores masto-citarios, y una fase tardía, algunas horas después, intensa-mente inflamatoria. Esta última se caracteriza por la infiltra-ción celular, principalmente de eosinófilos y linfocitos T, queson atraídos por factores quimiotácticos liberados por losmastocitos5 (fig. 1).La respuesta humoral implica, en primer lugar, el reconoci-miento del alergeno por parte de linfocitos Th específicos, yesto sólo puede ocurrir si dicho alergeno se encuentra aso-ciado físicamente a la molécula HLA en la superficie de lacélula presentadora de antígenos. El reconocimiento delalergeno induce en el linfocito T la secreción de sus linfoci-nas características, en particular IL-4. Posteriormente, la in-teracción del linfocito T con el linfocito B y la acción de laIL-4 sobre este último conducen a la síntesis de IgE.La IgE se produce en todos los individuos como parte de ladefensa natural frente a la infestación por parásitos helmin-tos6,7. Constituye una fracción minúscula en el total de anti-cuerpos del suero humano (50-300 ng/ml, frente a los 10mg/ml de la IgG), pero su acción se amplifica poderosa-mente por la actividad de los receptores a los que se une. Elreceptor de alta afinidad de la IgE (FcεRI, receptor para laregión constante de la IgE), presente en la superficie demastocitos y basófilos, es el causante de las reacciones in-mediatas de hipersensibilidad. Cuando un alergeno multiva-lente se asocia con los complejos FcεRI-IgE, se pone enmarcha la desgranulación de estas células: una rápida libe-ración de mediadores (histamina, leucotrienos, etc.), y lasíntesis y secreción de citocinas que atraen y activan a lascélulas inflamatorias8.
Factores implicados en el desarrollo de enfermedadesatópicas
Factores ambientales
Los factores ambientales pueden influir tanto sobre el co-mienzo como sobre la gravedad de la enfermedad atópica.Los más importantes en el desarrollo del asma alérgico sonla intensidad y la duración de la exposición a los aeroaler-genos. Los estudios epidemiológicos han postulado la rela-ción existente entre el grado de exposición a los alergenosy la sensibilización a los ácaros9 o los síntomas respiratoriosde alergia10. Según estos estudios, la exposición a más de2 µg del alergeno por gramo de polvo aumenta el riesgo desensibilización a los ácaros, mientras que la exposición a10 µg/g o más incrementa el riesgo de síntomas asmáti-cos11,12.

M.J. TORRES GALVÁN Y J.C. RODRÍGUEZ PÉREZ.– GENES Y FACTORES AMBIENTALES EN EL DESARROLLO DE ASMA Y ATOPIA
Fig. 1. Mecanismo de la reacción alérgica; CPA: célula presentadora de antígenos; FcεRI: receptor de alta afinidad de la IgE; IL: interleucina.
T B
Inflamación
Quimiotaxis
Antígeno
Presentación
IL-4
IgE Mediadores
FcεRI
CPA
Otros determinantes que en algunos estudios han demos-trado tener un papel en el desarrollo de alergias son la si-tuación socioeconómica, la condición atópica de la madre,el bajo peso al nacer, la estación de nacimiento y las infec-ciones de las vías respiratorias, principalmente virales13-16.
Factores genéticos
El componente hereditario de la respuesta IgE se detectó enlos primeros estudios17, al observarse un agrupamiento fa-miliar de las enfermedades atópicas. Hoy día no existe dudaalguna acerca de la contribución de los genes a la etiologíade las enfermedades alérgicas: los estudios familiares y engemelos han demostrado repetidamente la influencia de losgenes en el desarrollo de la atopia18,19.La atopia es una enfermedad de origen genético heterogé-neo, al igual que la hipertensión, la aterosclerosis y la diabe-tes. Todas estas enfermedades muestran un patrón heredi-tario que no puede ser clasificado de mendeliano simple, esdecir, no existe un único gen causante de la enfermedad,sino que hablamos de un conjunto de genes que confierensusceptibilidad al desarrollo de la enfermedad.
Métodos de identificación de genes relacionados con la enfermedad
Los genes que predisponen al individuo a la enfermedadpueden ser identificados más fácilmente a través del des-cubrimiento de una proteína anormal. Cuando la base bio-química o fisiológica de una enfermedad genética no se co-noce por completo, los genes pueden ser encontrados mediante dos estrategias básicas: la aproximación por ge-nes candidatos y la búsqueda en todo el genoma20.En la estrategia basada en genes candidatos, se comienzapor seleccionar aquéllos claramente implicados en el proce-so en cuestión, como es el caso de los HLA, receptor dellinfocito T, diversas citocinas y sus receptores, o el receptorde alta afinidad para la IgE, en la enfermedad atópica. El si-guiente paso consiste en la identificación en los genes esco-gidos de variantes alélicas que puedan estar asociadas a laenfermedad.
En la estrategia de búsqueda en todo el genoma, se utilizansecuencias de ADN polimórficas, principalmente microsaté-lites, que se encuentran a todo lo largo del mismo. Median-te análisis de ligamiento, se pueden identificar regiones cromosómicas que cosegregan con el fenotipo de la enfer-medad. Posteriormente se puede buscar el gen relacionadocon la enfermedad en la región concreta mediante clona-ción posicional, o estudiar directamente genes candidatoslocalizados en la zona de interés.A menudo se emplea una combinación de ambas estrate-gias. De hecho, con respecto al estudio de la atopia la apro-ximación más frecuente ha consistido en utilizar series demicrosatélites localizados en regiones cromosómicas congenes candidatos.
Herramientas: estudios de ligamiento y asociación
Los estudios de ligamiento están diseñados para investigarsi alelos concretos de un marcador se transmiten junto conla enfermedad más de lo esperado por azar. Se basan en larelación inversa que existe entre la distancia física entre dosgenes y la probabilidad de que ocurra una recombinaciónentre ambos durante la meiosis21. Un resultado de ligamien-to positivo indica, por tanto, que en la proximidad del mar-cador genético utilizado existe, al menos, un gen que contri-buyen a la enfermedad (fig. 2).Los estudios de asociación comparan las frecuencias de losalelos de un gen particular entre individuos enfermos y sa-nos. Este enfoque presupone una relación causal entre elgen y la enfermedad. Un alelo de un marcador y una enfer-medad están asociados si dicho alelo aparece en el grupode pacientes con una frecuencia significativamente alta22.
Base genética de la atopia
Tres son las principales regiones cromosómicas con genescandidatos que han obtenido resultados significativos en es-tudios de ligamiento o asociación con respecto a la atopia:la región HLA, la zona de la subunidad β del receptor dealta afinidad para la IgE y el agrupamiento de genes de cito-
699

MEDICINA CLÍNICA. VOL. 114. NÚM. 18. 2000
70
Fig. 2. Base genética de los estudios de ligamento. Y,y: alelos de un marca-dor genético; Z, z: alelos de un gen relacionado con la enfermedad, siendo Zel alelo que determina el fenotipo afecto.a. Si, en el cromosoma, el marcador está muy próximo al gen relacionadocon la enfermedad, no se produce recombinación entre ambos durante lameiosis, y el alelo Z se hereda junto con el alelo Y: los individuos afectadosson portadores del alelo Y.b. Si el marcador y el gen relacionado con la enfermedad están suficiente-mente alejados en el cromosoma, puede producirse recombinación entreambos y heredarse los alelos de todas las formas posibles: los individuosafectados pueden ser portadores del alelo Y o del y, indistintamente.
YZ
y z
Y Z
y z
YZ
y z
Y z
y Z
Y Z
y z
Progenitor Meiosis Gametos
Progenitor Meiosis Gametos
a
b
cinas del cromosoma 5. Las moléculas codificadas por es-tos genes tienen un papel clave en la reacción alérgica, co-mentado brevemente en un apartado anterior.
Región HLA: cromosoma 6
En el esfuerzo por comprender el papel de la respuesta in-munitaria en la patología que nos ocupa, se han realizadoestudios tanto de ligamiento como de asociación para exa-minar la relación entre la atopia y los genes HLA, localiza-dos en la región cromosómica 6p21.3.La influencia de HLA sobre la respuesta IgE fue observadapor primera vez en 1972, cuando Levine et al encontraronuna asociación entre haplotipos HLA y la respuesta al antí-geno E de Ambrosia artemisifolia23. Posteriormente se de-mostró la asociación de la respuesta al componente Amb aV del polen de dicha especie: en población blanca se obser-vó que la respuesta restringida por HLA se limitaba a los in-dividuos que presentaban el antígeno Dw224,25. Según susresultados, los individuos poseedores del Dw2 tenían unaprobabilidad 65 veces mayor de desarrollar una respuestaIgE frente al alergeno. Con el avance de las técnicas mole-culares se pudo establecer con posterioridad que las molé-culas causantes de esta asociación eran el HLA-DR*1501en blancos y el HLA-DR*1502 en asiáticos26,27.Se trata de la asociación más consistente encontrada hastael momento, pero no la única. Otros alergenos de los que sehan publicado asociaciones con alelos HLA son: Lol p I, II yIII (polen de Lolium perenne) y el DR328; Ole e I (polen delolivo) y el DRB1*07/DQB1*020129; Chi t I (hemoglobinasdel díptero Chironomus thummi) y DRB1*01/DQB1*0501/-DQA1*010130; Cry j I (polen de Cryptomeria japonica) y el
0
DP531. Todos estos alergenos tienen en común que estánbien caracterizados y se utilizan purificados en los respecti-vos estudios, lo que facilita los estudios de asociación.En cuanto a los ácaros (tabla 1), también existen trabajosque demuestran la existencia de ligamiento genético entrela respuesta a ellos y la región HLA32,33. Sin embargo, debi-do a que la fuerza de la asociación entre el HLA y los aler-genos disminuye a medida que aumenta la complejidadmolecular de éstos, no ha sido posible establecer asociacio-nes con alelos concretos en estudios epidemiológicos33-36,salvo en un caso en que se encontró una asociación delasma atópica por sensibilización a Der p y Der f con elDR*1137. No obstante, el tamaño muestral de este estudiono parece suficiente (20 pacientes), teniendo en cuentaque la prevalencia estimada del asma en la población vene-zolana es del 20%, según los propios autores.Los resultados de los estudios realizados indican que elefecto de los genes HLA es más fácilmente detectablecuando el antígeno es pequeño y posee un único, o unospocos, determinantes antigénicos, que es el caso de la ma-yoría de los alergenos mencionados anteriormente.En los últimos años, se ha descrito una nueva categoriza-ción de las moléculas HLA que se ha revelado útil en el es-tudio de ciertas enfermedades autoinmunes, como la artritisreumatoide y la diabetes tipo 1, ya que parece explicar con-tradicciones aparentes en los resultados de estudios simila-res38. Esta aproximación consiste en agrupar los alelos HLA-DBR1 según los aminoácidos que presentan la molécula enla zona de unión con el antígeno. De esta forma, se han de-finido 7 grupos, uno de los cuales ha demostrado tener unaasociación con la enfermedad atópica por sensibilización aDer p39, al reanalizar los datos HLA de un estudio previo enel que no se había encontrado asociación de ningún aleloHLA con la enfermedad atópica33. El grupo funcional encuestión estaba presente en el 44% de los pacientes, y losresultados del estudio sugieren que los individuos portado-res de un alelo HLA-DRB1 perteneciente a este grupo po-drían estar predispuestos a desarrollar una enfermedad ató-pica.Hasta la fecha se han identificado más de 200 alelos HLA-DRB1 distintos, según su secuencia en el ADN. Sin embar-go, las diferencias en la secuencia de aminoácidos de laproteína son mucho más importantes, ya que una únicasustitución en la zona de unión al antígeno puede alterar lacapacidad de la molécula para realizar dicha unión y, portanto, el signo de la respuesta inmunológica. Esta estrategiapodría facilitar el avance de los estudios que intentan en-contrar asociaciones significativas entre la respuesta a antí-genos complejos y los genes HLA, ya que los resultados delos estudios de asociación alélica habituales no parece quevayan a ser de utilidad clínica.
TABLA 1
Estudios de ligamiento y asociación entre los genes HLAy la respuesta a Dermatophagoides spp.
Autores Referencia Población Alergeno L A
Caraballo y 32 Mulatos Der f +Hernández, 1990 colombianos
Young et al, 1994 34 Británica Der p I – –Der p II – –
Holloway et al, 1996 35 Británica Der p –Der f –
Stephan et al, 1996 36 Alemana Der p I –Torres-Galván et al, 1999 33 Española Derp p + –Lara-Márquez et al, 1999 37 Mestizos Derp p, – DR*11
venezolanos Der fL: ligamiento; A: asociación; Der p: Dermatophagoides pteronyssinus; Der f: Dermatopha-goides farinae.

M.J. TORRES GALVÁN Y J.C. RODRÍGUEZ PÉREZ.– GENES Y FACTORES AMBIENTALES EN EL DESARROLLO DE ASMA Y ATOPIA
Subunidad β del receptor de alta afinidad para la IgE:cromosoma 11
La región cromosómica 11q13 fue la segunda implicada enla patogenia de la atopia, a raíz de un análisis de ligamientoen el que se utilizaron 17 marcadores genéticos distribuidospor todo el genoma40. Posteriormente se localizó el gen quecodifica la cadena β del receptor de alta afinidad para la IgE(FcεRI-β) en dicha zona41.Estos resultados generaron un interés considerable, y diver-sos grupos han intentado desde entonces encontrar eviden-cias de ligamiento con diversos marcadores localizados en11q13. Ha habido algunos resultados positivos en poblacióneuropea42-44 y australiana45,46. Sin embargo, otros investiga-dores de Japón47, Australia48, Europa49,50 y los Estados Uni-dos51,52 no han podido confirmar el ligamiento en sus pobla-ciones. En población española se han realizado dosestudios de asociación con sendos marcadores intrónicosdel gen FcεRI-β, con resultados negativos en ambos ca-sos53,54.En los últimos años, las investigaciones en torno al genFcεRI-β han dado lugar a la identificación de varios polimor-fismos exónicos, desde que Shirakawa et al55 describieranuna variante del mismo (Ile181Leu, sustitución de una iso-leucina por una leucina en la posición 181 de la proteína)que cosegregaba con la atopia. Algunos de estos polimorfis-mos han sido asociados positivamente con distintas carac-terísticas del fenotipo atópico como la IgE elevada56, la hipe-rreactividad bronquial57-59 o el asma59-61 en distintaspoblaciones. Nuevamente aparecen estudios que no hanproporcionado evidencia de esta relación54,61-63.
Agrupamiento de genes de citocinas: cromosoma 5
El agrupamiento de genes de citocinas implicadas en la re-acción alérgica, localizado en la región 5q31-q33, ha hechode esta zona una buena candidata a contener un «gen ató-pico». Aquí se encuentran los genes de la IL-3, IL-4, IL-5,IL-9, IL-13, y del factor de crecimiento de granulocitos ymacrófagos (GM-CSF), entre otros.El gen de la IL-4 se ha estudiado insistentemente desdeque se describió un ligamiento del mismo con títulos eleva-dos de IgE total64. También se ha obtenido evidencia de li-gamiento e, incluso, de asociación alélica en otros trabajosrealizados en población europea45,46, australiana65 y japone-sa66, aunque en otros casos no se ha podido llegar a lasmismas conclusiones67-69. Recientemente, un estudio multi-céntrico realizado en los Estados Unidos sobre tres gruposétnicos (afroamericanos, blancos e hispanos) ha obtenidouna significación modesta de ligamiento de la región 5q31con el asma en población blanca, y los autores apuntan queesta zona podría tener una importancia mayor para otros fe-notipos asociados con el asma como la IgE o la hiperreacti-vidad bronquial53. Un estudio realizado recientemente enpoblación española ha tenido como resultado la falta deasociación de un marcador intrónico del gen de la IL-4 conla enfermedad atópica por sensibilización a Der p54.En 1995, un estudio realizado en familias estadounidensescon asma reveló una asociación entre un polimorfismo delpromotor de la IL-4 (C-590T, sustitución de una citosina poruna timina en la posición-590) y títulos altos de IgE total70.El análisis de la expresión del gen puso en evidencia unamayor transcripción de la proteína en los individuos que po-seían dicha variante, coincidiendo con los datos de IgE. Enrelación con la atopia y el asma, este polimorfismo demostróuna asociación débil con la IgE específica a Der p en pobla-ción australiana71. También se ha publicado una asociacióncon el asma en niños japoneses, aunque los autores indican
que esta relación no se establece a través de la modulaciónde la IgE total72. Un estudio reciente en población europeaexcluye la contribución de esta variante a la regulación delos valores de IgE73.
Otros genes
Aparte de los mencionados anteriormente, existe una serieextensa de genes que están siendo estudiados en relacióncon la susceptibilidad para el desarrollo de alergias. Susproductos actúan en puntos diversos de esta compleja tra-ma, e incluyen no sólo proteínas implicadas en el reconoci-miento o distintos tipos de citocinas, sino también factoresde transcripción, receptores y moléculas de adhesión. Algu-nos ejemplos son el receptor adrenérgico β2 (cromosoma5)74,75, el factor de necrosis tumoral (región HLA)76,77 o el re-ceptor de la IL-4 (cromosoma 16)78,79.
Conclusiones
La atopia es uno de los principales factores de riesgo en eldesarrollo de asma. En los individuos con asma atópica, lainhalación del alergeno produce una respuesta bifásicacuya característica clínica es la obstrucción reversible de lasvías respiratorias. Dado que el asma y otras enfermedadesatópicas tienen una base genética, es razonable esperarque su conocimiento conduzca a mejoras en el diagnósticoy tratamiento de estas afecciones. Además, la detecciónprecoz de individuos genéticamente predispuestos al desa-rrollo de enfermedades atópicas permitirá la aplicación demedidas preventivas con lo que disminuiría la incidencia de una afección que comporta un coste socioeconómicoconsiderable.En los últimos años, se ha realizado una gran cantidad deestudios dirigidos a identificar factores genéticos de predis-posición, con resultados desiguales. Hasta el momento noha sido posible, salvo en algún caso muy concreto, estable-cer relaciones directas entre genes y enfermedades alérgi-cas que pudieran ser de utilidad clínica. La complejidad dela trama celular y molecular de la reacción alérgica, que setraduce en una gran heterogeneidad genética de la enfer-medad, sin duda dificulta estas investigaciones, junto conlas posibles diferencias étnicas y metodológicas.A pesar de las limitaciones metodológicas inherentes a losestudios genéticos de enfermedades complejas, se está pro-gresando en la identificación de regiones cromosómicasque influyen en el fenotipo atópico. Confirmar los resultadospositivos en muestras independientes, analizar las interac-ciones entre los distintos genes implicados, y con los fac-tores ambientales, y dilucidar el verdadero mecanismo deestas enfermedades son los principales retos para los próxi-mos años.
REFERENCIAS BIBLIOGRÁFICAS
1. The International Study of Asthma and Allergies in Childhood (ISAAC)Steering Committee. Worldwide variation in prevalence of symptoms ofasthma, allergic rhinoconjunctivitis, and atopic eczema: ISAAC. Lancet1998; 351: 1225-1232.
2. Cookson W. The alliance of genes and environment in asthma andallergy. Nature (Lond) 1999; 402 (Supl 6760): B5-11.
3. Von Pirquet C. Allergie. Münch Med Wochenschr 1906; 30: 1457.4. Coca AF, Cooke RA. On the phenomenon of hypertensitiveness. J Immu-
nol 1923; 8: 163-182.5. Kapsenberg ML, Hilkens CMU, Jansen HM, Bos JD, Snijders A, Wieren-
ga EA. Production and modulation of T-cell cytokines in atopic allergy.Int Arch Allergy Immunol 1996; 110: 107-113.
6. Maizels RM, Bundy DAP, Selkirk ME, Smith DF, Anderson RM. Immu-nological modulation and evasion by helminth parasites in human popu-lations. Nature (Lond) 1993; 365: 797-805.
701

MEDICINA CLÍNICA. VOL. 114. NÚM. 18. 2000
7
7. Gounni AS, Lamkhioued B, Ochial K, Tanaka Y, Delaporte E, Capron Aet al. High-affinity IgE receptor on eosinophils is involved in defenceagainst parasites. Nature (Lond) 1994; 367: 183-186.
8. Baraniuk JN. Pathogenesis of allergic rhinitis. J Allergy Clin Immunol1997; 99: S763-772.
9. Young RP, Hart BJ, Merrett TG, Read AF, Hopkin JM. House dust mitesensitivity: interaction of genetics and allergen dosage. Clin Exp Allergy1992; 22: 205-211.
10. Sporik R, Holgate ST, Platts-Mills TA, Cogswell JJ. Exposure to house-dust mite allergen (Der p I) and the development of asthma in childho-od. A prospective study. N Engl J Med 1990; 323: 502-507.
11. Lau S, Falkenhorst G, Weber A, Werthmann I, Lind P, Buettner-Goetz P etal. High mite-allergen exposure increases the risk of sensitization in atopicchildren and young adults. J Allergy Clin Immunol 1989; 84: 718-725.
12. Sporik R, Chapman MD, Platts-Mills TAE. House dust mine exposure asa cause of asthma. Clin Exp Allergy 1992; 22: 897-906.
13. Arshad SH, Stevens M, Hide DW. The effect of genetic and environmen-tal factors on the prevalence of allergic disorders at the age of two years.Clin Exp Allergy 1993; 23: 504-511.
14. Bonini S, Magrini L, Rotiroti G, Ronchetti MP, Onorati P. Genetic and en-vironmental factors in the changing incidence of allergy. Allergy 1994;49: 6-14.
15. Omenaas E, Bakke P, Elsayed S, Hanoa R, Gulsvik A. Total and specificserum IgE in adults: relationship to sex, age and environmental factors.Clin Exp Allergy 1994; 24: 530-539.
16. Sandford A, Weir T, Paré P. The genetics of asthma. Am J Respir CritCare Med 1996; 153: 1749-1765.
17. Cooke RA, Van der Veer A Jr. Human sensitization. J Immunol 1916; 1:201-305.
18. Blumenthal MN, Namboodiri K, Mendell N, Gleich G, Elston RC, YunisE. Genetic transmission of serum IgE levels. Am J Med Genet 1981; 10:219-228.
19. Hanson B, McGue M, Roitman-Johnson B, Segal NL, Bouchard TJ Jr,Blumenthal MN. Atopic disease and immunoglobulin E in twins rearedapart and together. Am J Hum Genet 1991; 48: 873-879.
20. Ober C. Genetics of atopy. En: Barnes PJ, Grusntein MM, Leff AR, Wool-cock AJ, editores. Asthma: genetics of atopy. Filadelfia: Lippincott-RavenPublishers, 1997; 129-144.
21. Ruffilli A, Bonini S. Susceptibility genes for allergy and asthma. Allergy1997; 52: 256-273.
22. Elston RC. Linkage and association to genetic markers. Exp Clin Immu-nogenet 1995; 12: 129-140.
23. Levine BB, Stember RH, Fontino M. Ragweed hayfever: genetic controland linkage to HL-A haplotypes. Science 1972; 178: 1201-1203.
24. Marsh DG, Hsu SH, Roebber M, Ehrlich-Kautzky E, Freidhoff LR, MeyersDA et al. HLA-Dw2: a genetic marker for human immune response toshort ragweed pollen allergen Ra5. I. Response resulting primarily fromnatural antigenic exposure. J Exp Med 1982; 155: 1439-1451.
25. Marsh DG, Meyers DA, Freidhoff LR, Ehrlich-Kautzky E, Roebber M,Norman PS et al. HLA-Dw2: a genetic marker for human immune res-ponse to short ragweed pollen allergen Ra5. II. Response after ragweedimmunotherapy. J Exp Med 1982; 155: 1452-1463.
26. Huang SK, Zwollo P, Marsch DG. Class I major histocompatibility com-plex restriction of human T cell responses to short ragweed allergen,Amb a V. Eur J Immunol 1991; 21: 1469-1473.
27. Zwollo P, Ehrlich-Kautzky E, Scharf SJ, Ansari AA, Erlich HA, Marsh DG.Sequencing of HLA-D in responders and non-responders to short rag-weed allergen, Amb a V. Immunogenetics 1991; 33: 141-151.
28. Freidhoff LR, Ehrlich E, Meyers DA, Ansari AA, Bias WB, Marsh DG. As-sociation of HLA-DR3 with human immune response to Lo1 p I and Lo1p II alergens in allergic subjects. Tissue Antigens 1988; 31: 211-219.
29. Cárdaba B, Vilches C, Martín E, De Andrés B, Del Pozo V, Hernández Det al. DR7 and DQ2 are positively associated with immunoglobulin-E res-ponse to the main antigen of olive pollen (Ole e I) in allergic patients.Hum Immunol 1993; 38: 293-299.
30. Tautz C, Rihs HP, Thiele A, Zwollo P, Freidhoff LR, Marsh DG et al. Associaton of class II sequences encoding DR1 and DQ5 specificitieswith hypersensitivity to chironomid allergen Chit I. J Allergy Clin Immunol1994; 93: 918-925.
31. Hori T, Kamikawaji N, Kimura A, Sone T, Komiyama N, Komiyama S etal. Japanese cedar pollinosis and HLA-DP5. Tissue Antigens 1996; 47:485-491.
32. Caraballo LR, Hernández M. HLA haplotype segregation in families withallergic asthma. Tissue Antigens 1990; 35: 182-186.
33. Torres-Galván MJ, Quiralte J, Blanco C, Castillo R, Carrillo T, Pérez-Acie-go P et al. Linkage of house dust mite allergy with the HLA region. AnnAllergy Asthma Immunol 1999; 82: 198-203.
34. Young RP, Dekker JW, Wordsworth BP, Schou C, Pile KD, Matthiesen Fet al. HLA-DR and HLA-DP genotypes and immunoglobulin E responsesto common major allergens. Clin Exp Allergy 1994; 24: 431-439.
35. Holloway JW, Doull I, Begishvili B, Beasley R, Holgate ST, Howell WM.Lack of evidence of a significant association between HLA-DR, DQ andDP genotypes and atopy in families with HDM allergy. Clin Exp Allergy1996; 26: 1142-1149.
36. Stephan V, Schmid V, Frischer T, Sparholt S, Forster J, Whan V et al.Mite allergy, clinical atopy, and restriction by HLA class II immune res-ponse genes. Pediatr Allergy Immunol 1996; 7: 28-34.
02
37. Lara-Márquez ML, Yunis JJ, Layrisse Z, Ortega F, Carvallo-Gil E, Montag-nani S et al. Immunogenetics of atopic asthma: association ofDRB1*1101 DQA1*0501 DQB1*0301 haplotype with Dermatophagoi-des spp.-sensitive asthma in a sample of the Venezuelan population.Clin Exp Allergy 1999; 29: 60-71.
38. Ou D, Mitchell LA, Tingle AJ. A new categorization of HLA-DR alleles ona functional basis. Hum Immunol 1998; 59: 665-676.
39. Torres-Galván MJ, Quiralte J, Blanco C, Castillo R, Carrillo T, Pérez-Acie-go P et al. Pocket 4 in the HLA-DRB1 binding groove: an associationwith atopy. Allergy 2000; 55: 398-401.
40. Cookson WO, Sharp PA, Faux JA, Hopkin JM. Linkage between immu-noglobulin E responses underlying asthma and rhinitis and chromosome11q. Lancet 1989; 1: 1292-1295.
41. Sandford AJ, Shirakawa T, Moffatt MF, Daniels SE, Ra C, Faux JA et al.Localization of atopy and β subunit of high-affinity IgE receptor (FcεRI)on chromosome 11q. Lancet 1993; 341: 332-334.
42. Collée JM, Kate LP, De Vries HG, Kliphuis JW, Bouman K, Scheffer H etal. Allele sharing on chromosome 11q13 in sibs with asthma and atopy.Lancet 1993; 342: 936.
43. Doull IJ, Lawrence S, Watson M, Begishvili T, Beasley RW, Lampe F etal. Allelic association of gene markers on chromosomes 5q and 11q withatopy and bronchial hyperresponsiveness. Am J Respir Crit Care Med1996; 153: 1280-1284.
44. Palmer LJ, Daniels SE, Rye PJ, Gibson NA, Tay GK, Cookson WO et al.Linkage of chromosome 5q and 11q gene markers to asthma associatedquantitative traits in Australian children. Am J Respir Crit Care Med1998; 158: 1825-1830.
45. Van Herwerden L, Harrap SB, Wong ZYH, Abramson MJ, Kutin JJ, For-bes AB et al. Linkage of high affinity-IgE receptor gene with bronchial hy-perreactivity, even in absence of atopy. Lancet 1995; 346: 1262-1265.
46. Daniels SE, Bhattacharyya S, James A, Leaves NI, Young A, Hill MR etal. A genome-wide search for quantitative trait loci underlying asthma.Nature (Lond) 1996; 383: 247-250.
47. Hizawa N, Yamaguchi E, Ohe M, Itoh A, Furuya K, Ohnuma N et al.Lack of linkage between atopy and locus 11q13. Clin Exp Allergy 1992;22: 1065-1069.
48. Brereton HM, Ruffin RE, Thompson PJ, Turner DR. Familial atopy inAustralian pedigrees: adventious linkage to chromosome 8 is not confir-med nor is there evidence of linkage to the high affinity IgE receptor. ClinExp Allergy 1994, 24: 868-877.
49. Coleman R, Trembath RC, Harper JI. Chromosome 11q13 and atopyunderlying atopic eczema. Lancet 1993; 341: 1121-1122.
50. Deichmann KA, Starke B, Schlenther S, Heinzmann A, Sparholt SH,Forster J et al. Linkage and association studies of atopy and the chromo-some 11q13 region. J Med Genet 1999; 36: 379-382.
51. The Collaborative Study on the Genetics of Asthma (CSGA). A genome-wide search for asthma susceptibility loci in ethnically diverse popula-tions. Nat Genet 1997; 15: 389-392.
52. Amelung PJ, Postma DS, Xu J, Meyers DA, Bleecker ER. Exclusion ofchromosome 11q and the FcεRI-β gene as aetiological factors in allergyand asthma. in a population of Dutch asthmatics families. Clin ExpAllergy 1998; 28: 397-403.
53. Castro J, Telleria JJ, Blanco-Quiros A, Linares P, Andion R. Lack of asso-ciation between atopy and RsaI polymorphism within intron 2 of theFc(epsilon)RI-beta gene in a Spanish population sample. Allergy 1998;53: 1083-1086.
54. Torres Galván MJ. Factores genéticos implicados en el desarrollo de asma yrinoconjuntivitis por sensibilización al ácaro Dermatophagoides pteronyssi-nus en la población de las Islas Canarias. Universidad de Las Palmas deGran Canaria (ULPGC) [tesis doctoral]. Las Palmas de Gran Canaria, 1999.
55. Shirakawa T, Li A, Dubowitz M, Dekker JW, Shaw AE, Faux JA et al. As-sociation between atopy and variants of the beta subunits of the high-affinity immunoglobulin E receptor. Nat Genet 1994; 7: 125-129.
56. Paré PD, Faux JA, Hill MR, Kan R, Bremmer P, Musk M et al. The pre-valence of the Leu 181/183 polymorphism in the gene for the high affi-nity receptor (FcεRI-β) in Australian aborigines: association with atopy[resumen]. Am J Respir Crit Care Med 1995; 151: A214.
57. Hill MR, James AL, Faux JA, Ryan G, Hopkin JM, Le Souef P et al. Fcε−RI-β polymorphism and risk of atopy in a general population sample. BrMed J 1995; 311: 776-779.
58. Hill MR, Cookson W. ocm. A new variant of the β subunit of the high-affinityreceptor for immunoglobulin E (FcεRI-β E237G): associations with measu-res of atopy and bronchial hyper-responsiveness. Hum Mol Genet 1996.
59. Martinati LC, Malerba G, Cusin V, Boner AL, Pignatti PF, Trabetti E. As-sociation of two DNA polymorphisms in the FCεRIβ gene with bronchialhyperresponsiveness in an Italian population. Eur Respir J 1997; 10(Supl 25): 302S.
60. Hijazi Z, Haider MZ, Khan MR, Al-Dowaisan AA. High frequency of IgEreceptor FcεRI-β variant (Leu181/Leu183) in Kuwaiti Arabs and its asso-ciation with asthma. Clin Genet 1998; 53: 149-152.
61. Duffy DL, Healey SC, Chenevix-Trench G, Martin NG, Weger J, Lichter J.Atopy in Australia. Nat Genet 1995; 10: 260.
62. Fukao T, Kaneko H, Teramoto T, Tashita H, Kondo N. Association be-tween FcεRI-β and atopic disorder in Japanese population? Lancet1996; 348: 407.
63. Hall IP, Wheatley A, Dewar J. FcεRI-β polymorphisms unlikely contributesubstantially to genetic risk of allergic disease. Br Med J 1996; 312: 311.

M.J. TORRES GALVÁN Y J.C. RODRÍGUEZ PÉREZ.– GENES Y FACTORES AMBIENTALES EN EL DESARROLLO DE ASMA Y ATOPIA
64. Marsh DG, Neely JD, Breazeale DR, Ghosh B, Freidhoff LR, Ehrlich-Kautzky E et al. Linkage analysis of IL-4 and other chromosome 5q31.1markers and total serum immunoglobulin E concentrations. Science1994; 264: 1152-1156.
65. Walley AJ, Bhattacharyya S, Leaves N, Daniels SE, Cookson WOCM. Lin-kage and allelic association of chromosome 5 microsatellite markers withatopic asthma phenotypes in a general population sample. Am RespirCrit Care Med 1997; 155: A257.
66. Noguchi E, Shibasaki M, Arinami T, Takeda K, Maki T, Miyamoto T et al.Evidence for linkage between asthma/atopy in childhood and chromoso-me 5q31-q33 in a Japanese population. Am J Respir Crit Care Med1997; 156: 1390-1393.
67. Blumenthal MN, Wang Z, Weber JL, Rich SS. Absence of linkage be-tween 5q markers and serum IgE levels in four large atopic families. ClinExp Allergy 1996; 26: 892-896.
68. Ulbrecht M, Eisenhut T, Bönisch J, Kruse R, Wjst M, Heinrich J et al.High serum IgE concentrations: association with HLA-DR and markerson chromosome 5q31 and chromosome 11q13. J Allergy Clin Immunol1997; 99: 828-836.
69. Mansur AH, Bishop DT, Markham AF, Britton J, Morrison JFJ. Associa-tion study of asthma and atopy traits and chromosome 5q cytokine clus-ter markers. Clin Exp Allergy 1998; 28: 141-150.
70. Rosenwasser LJ, Klemm DJ, Dresback JK, Inamura H, Mascali JJ, Klin-nert M et al. Promoter polymorphisms in the chromosome 5 gene clusterin asthma and atopy. Clin Exp Allergy 1995; 25 (Supl 2): 74-78.
71. Walley AJ, Cookson WOCM. Investigation of an interleukin-4 promoterpolymorphism for associations with asthma and atopy. J Med Genet1996; 33: 689-692.
72. Noguchi E, Shibasaki M, Arinami T, Takeda K, Yokouchi Y, KawashimaT et al. Association of asthma and the interleukin-4 promoter gene in Ja-panese. Clin Exp Allergy 1998; 28: 449-453.
73. Dizier MH, Sandford A, Walley A, Philippi A, Cookson W, Demenais F.Indication of linkage of serum IgE levels to the interleukin-4 gene and ex-clusion of the contribution of the (–590 C to T) interleukin-4 promoterpolymorphism to IgE variation. Genet Epidemiol 1999; 16: 84-94.
74. Dewar JC; Wilkinson J, Wheatley A, Thomas NS, Doull I, Morton N et al.The glutamine 27 β2-adrenoceptor polymorphism is associated with ele-vated IgE levels in asthmatics families. J Allergy Clin Immunol 1997;100: 61-65.
75. Deichmann KA, Schmidt A, Heinzmann A, Kruse S, Forster J, Kuehr J.Association studies of beta2-adrenoceptor polymorphisms and enhan-ced IgE responsiveness in an atopic population. Clin Exp Allergy 1999;29: 794-799.
76. Moffatt MF, Cookson WOCM. Tumour necrosis factor haplotypes andasthma. Hum Mol Genet 1997; 6: 551-554.
77. Li Kam Wa TC, Mansur AH, Britton J, Williams G, Pavord I, Richards Ket al. Association between -308 tumour necrosis factor promoter poly-morphism and bronchial hyperreactivity in asthma. Clin Exp Allergy1999; 29: 1204-1208.
78. Kruse S, Japha T, Tedner M, Sparholt SH, Forster J, Kuehr J et al. Thepolymorphisms S503P and Q576R in the interleukin-4 receptor alphagene are associated with atopy and influence the signal transduction.Immunology 1999; 96: 365-371.
79. Rosa-Rosa L, Zimmermann N, Bernstein JA, Rothenberg ME, KhuranaHershey GK. The R576 IL-4 receptor alpha allele correlates with asthmaseverity. J Allergy Clin Immunol 1999; 104: 1008-1014.
703